La lutte contre le dopage est vraiment au cœur de tous les états qui souhaitent promouvoir le sport « propre ».
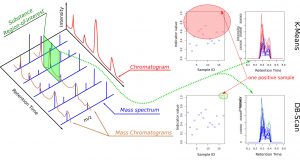
La méthode usuelle pour le contrôle antidopage est la chromatographie liquide associée à la spectrométrie de masse (LC-MS) sur les fluides des athlètes (urine, sang). L’ensemble des temps de rétention des substances chimiques (chromatographie liquide) et de leurs masses (spectrométrie de masse) sont scannés, ce qui conduit à l’acquisition de quantités élevées de données – entre 0.5 Go et 1 Go par athlète. Ainsi, pour chaque athlète deux spectres sont obtenus (spectre des ions positifs et spectre des ions négatifs) en trois dimensions (temps de rétention, masse m/z et intensité) (Figure 1, volet de gauche). Chaque substance se trouve donc dans une petite région d’intérêt d’un des deux spectres et centrée à des coordonnées propres (temps de rétention, masse) aux dérives expérimentales près. Ces régions d’intérêt sont comme petites fenêtres associées à chacune des plus de 600 substances à identifier, comme par exemple celle illustrée en vert dans le spectre représenté en Figure 1.
L’Agence Française de Lutte contre le Dopage (AFLD) effectue traditionnellement un contrôle visuel qui consiste à comparer les amplitudes des pics situés aux coordonnées attendues pour chacune des substances avec des seuils connus et caractérisés à l’avance par le laboratoire, la réponse est donc binaire : positive/négative à la présence d’un dopant. Cette étape est longue et requiert une grande connaissance des substances et du métabolisme humain. D’une manière générale, cette méthode d’analyse par comparaison de seuil, qui donne un résultat binaire, ne permet pas de mettre facilement en évidence des profils suspects d’athlètes dopés mais apparaissant en dessous des seuils ; pour ces situations, de véritables enquêtes seraient nécessaires.
Le département des analyses de l’AFLD, le CEA et le CNRS ont uni leurs forces pour automatiser la détection de substances prohibées dans les fluides des athlètes et ainsi limiter le risque de faux négatifs ou de faux positifs mais aussi pour détecter des profils d’athlètes suspects négatifs aux tests.
Comme il s’agit tout simplement d’un problème de classification, nous avons choisi des mettre en œuvre des algorithmes qui génèrent une information à trois états : positifs/négatifs/douteux. En production, cette approche ne nécessite pas d’intervention de la part d’un expert, sauf pour la validation finale qui est réglementaire. Nous avons travaillé sur l’intégralité du spectre en trois dimensions des échantillons réels fournis par l’AFLD et le volume de données est donc conséquent. Aussi avons-nous dû mettre au point un système de gestion de base de données permettant d’enregistrer les spectres puis d’y accéder via de simples requêtes SQL.
A priori, la plupart, des échantillons issus de contrôles anti-dopage sont négatifs aux substances prohibées car, d’une part de nombreuses substances de la base de données ne sont plus utilisées, et d’autre part les athlètes sont devenus extrêmement vigilants. Il n’est donc pas possible de les exploiter pour tester un classifieur, qui est un outil de traitement de donnée permettant d’automatiquement étiqueter des données, ou pour évaluer les performances d’une approche. Un générateur a donc été mis au point afin de rendre artificiellement positifs des échantillons et ainsi permettre de travailler avec des cohortes d’athlètes virtuels qui auraient pu être dopés à des substances même improbables et ce en respectant les spécificités de la spectrométrie de masse.
Les méthodes de classification sur des gros volumes de données nécessitent d’importants moyens de calcul. Pour cette raison plutôt que d’injecter des spectres entiers de près d’un giga-octets, beaucoup trop volumineux, dans un ou des classifieurs, nous avons défini un indicateur mathématique (Eq. 1) dont les valeurs servent de données d’entrée aux classifieurs. Il est défini comme le rapport entre la moyenne d’une zone d’intérêt du spectre pour une substance considérée et la médiane de la zone d’intérêt de la totalité des spectres associés à un événement sportif. Cela permet d’obtenir des résultats de classification rapidement puisque les vecteurs de données sont simplifiés à l’extrême.
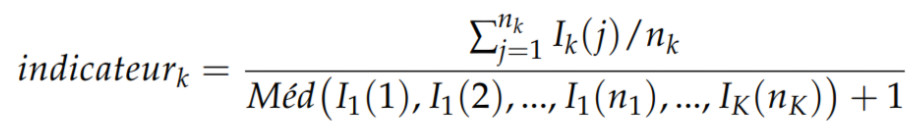
Plusieurs méthodes de classification ont été envisagées et nous avons retenu, une combinaison formée de K-Means et de DB-Scans (Figure 1, volet de droite) car d’une part ils ne nécessitent pas d’apprentissage à proprement parler, et d’autre part ils permettent de regrouper les données en différentes classes. Le premier va forcer la création de K classes tandis que l’autre se limitera à deux. Les sportifs pour lesquels le ou les indicateurs se trouvent trop éloignés de ceux des autres sportifs sont identifiés comme suspect. En outre, il s’agit d’outils classiques disponibles dans plusieurs langages de programmation, ce qui facilite un éventuel portage ou transfert de la proposition. Comme l’approche que nous proposons recherche l’anormalité des indicateurs (ceux qui sortent du lot) afin de détecter les cas douteux, alors un biais est possible si la totalité des athlètes est dopée de manière uniforme à la même substance. Pour éviter ce biais nous avons étendu l’approche en mixant les spectres associés à différents athlètes de différentes épreuves. Cela réduit à quasiment nulle cette éventualité ce que nos tests sur données synthétique ont confirmés
La méthode a été testée sur les spectres réels de six campagnes d’une trentaine d’athlètes chacune. Les résultats obtenus sont conformes à la méthode traditionnelle pour les athlètes dopés, avec en plus l’identification de profils suspects. Un schéma spécifique de base de données a été conçu, qui intéresse d’ailleurs d’autres collègues utilisant la spectrométrie de masse comme par exemple le laboratoire d’analyse des métabolites ou bien la recherche de pesticides dans les denrées alimentaires. L’algorithme exploite un indicateur mathématique ainsi que des algorithmes de classification. Les dérives expérimentales sont prises en compte afin de pouvoir combiner des spectres issus de compétitions différentes. La technique a été transférée auprès de la société Mesures-AD qui proposera un service en ligne d’ici quelques mois avec l’AFLD comme bêta-testeur pressenti ; elle pourra y déposer ses spectres anonymes à tester.
Contacts :
- Mathieu Thévenin, Université Paris-Saclay, UMR SPEC CEA-CNRS, Centre CEA-Saclay
- Paul Malfrait, CEA, actuellement en thèse à l’IRSN
- Robert French, Université de Bourgogne, Laboratoire d'Etude de l'Apprentissage et du Développement LEAD – CNRS UMR 5022.