L’ADN est une molécule très longue qui se retrouve compactée au sein des cellules. Ceci est permis grâce à l’association avec des protéines qui assurent la stabilité, la compaction et la régulation du matériel génétique. Les cellules eucaryotes et procaryotes s’appuient ainsi sur des assemblages macromoléculaires pour assurer une compaction optimale de l’ADN dans la cellule.
La présente étude met en lumière un mécanisme unique dans lequel une protéine amyloïde façonne l’ADN en s’oligomérisant autour de l’ADN, plutôt que d’enrouler l’ADN autour d’une protéine, comme cela est observé dans les nucléosomes eucaryotes. Ces résultats redéfinissent le paysage fonctionnel des amyloïdes, les liant à l’architecture du génome et à la régulation des gènes. Ils suggèrent que les interactions amyloïde-ADN pourraient représenter un mécanisme conservé à travers les systèmes biologiques, avec des implications importantes pour la compréhension de l’organisation du génome et la régulation de l’expression des gènes tant chez les procaryotes que chez les eucaryotes.
Cette étude portée par une équipe du LLB/IRAMIS, associe plusieurs équipes françaises (Synchrotron SOLEIL, ILL et UGA Grenoble, Université Paris Cité) et internationales (Espagne, Japon, Singapour, Royaume uni et Danemark).
Tous les organismes dépendent de protéines spécifiques pour compacter et organiser leur génome au sein de leurs cellules. Chez les eucaryotes (cellules avec noyau), les histones (protéines) remplissent ce rôle, tandis que les chromosomes bactériens des procaryotes (sans noyau) sont façonnés par des protéines dénommées– NAPS (pour Nucleoid Associated Protein).
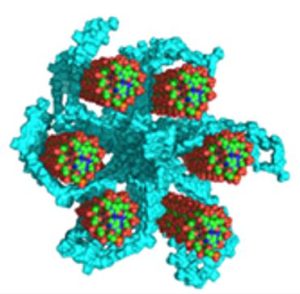
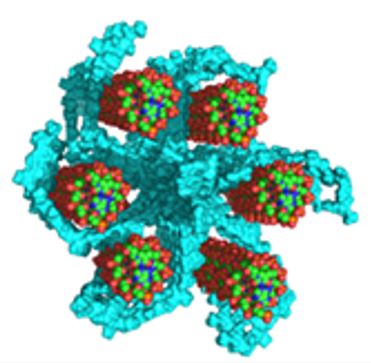
Parmi ces NAPs, la protéine Hfq joue un rôle central dans l’organisation du génome bactérien [1]. En utilisant une approche intégrative combinant diverses techniques de biophysique, dont la cryo-microscopie électronique à transmission, la diffusion de neutrons, la mutagenèse dirigée et la modélisation moléculaire, il a pu être identifié que le module amyloïde formé par la région C-terminale de Hfq compacte de manière inédite six molécules d’ADN, constituant la première description d’une structure amyloïde permettant la structuration de l’ADN.
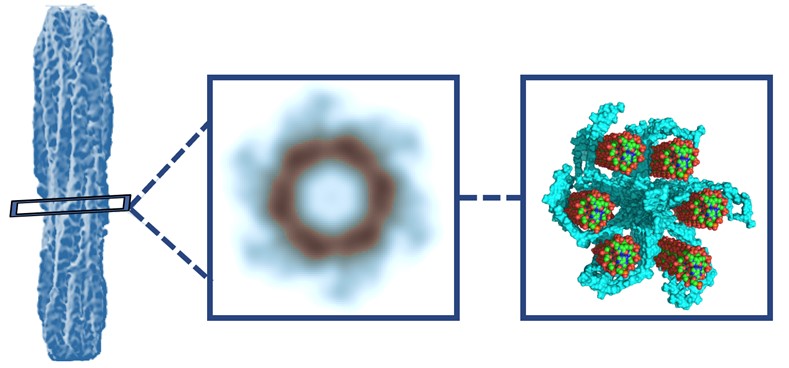
Structure transverse à symétrie hexagonale d’une fibre Hfq amyloïde-ADN compactée.
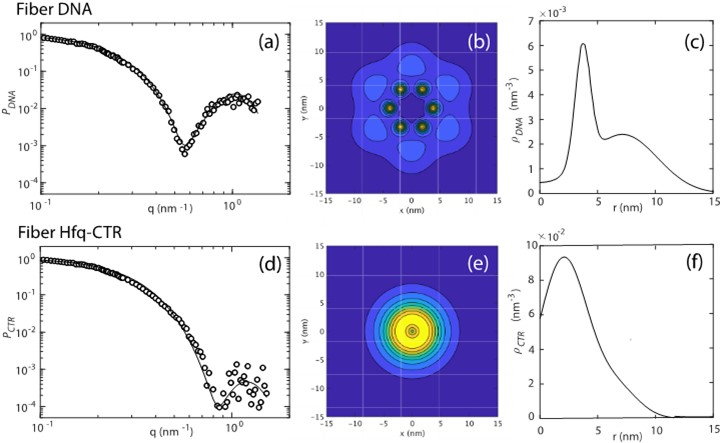
L’étude par diffusion des neutrons aux petits angles (SANS) a été réalisée à l’aide du diffractomètre aux petits angles à grande dynamique D22 à l’Institut Laue Langevin (ILL) à Grenoble (France) : une série d’échantillons a été préparée en dissolvant ces fibres amyloïdes dans des mélanges de H2O et D2O à une concentration d’ADN et de Hfq-CTR de 15 et 3,75 g/l. Pour l’étude, une longueur d’onde de 0,6 nm a été sélectionnée et la distance échantillon-détecteur mobile varie de 1,4 m à 17,6 m. Le temps de comptage total pour toutes les configurations de détecteur est de ∼2 h par échantillon. Ces expériences donnent des indications fortes sur les arrangements entre la protéine Hfq et l’ADN et montre notamment son rôle dans le pontage des molécules d’ADN. Complété par des images de microscopie électronique à transmission (Cryo-TEM), on obtient une idée précise de la conformation en six molécules d’ADN compactées ensemble par les protéines Hfq. Certaines bifurcations sont observées dans la reconstruction cryo-EM, qui pourraient résulter de la dissociation de l’ADN en brins simples induite par Hfq.
La diffusion inélastique des neutrons a alors été utilisée pour mesurer les vibrations moléculaires qui révèlent des détails sur les formes des potentiels d’interaction au sein des fibres. Cette technique permet une étude directe des changements de conformation des protéines, car les mouvements de grande amplitude impliqués dans le processus se situent dans la bonne gamme de fréquences mesurée par la technique. La dynamique des glaces cristallines D2O Ih (tampon), D2O + ADN, D2O + Hfq et D2O + Hfq + ADN a été étudiée à la température de 10 K, avec le spectromètre TOSCA auprès de la source de neutrons ISIS du laboratoire Rutherford Appleton (Royaume-Uni). Les changements spectraux confirment l’hypothèse de l’ouverture partielle de l’ADN et un effet du Hfq sur le pontage du désoxyribose. De plus un mode vibrationnel fort à 1625 cm-1 montre des interactions entre Hfq et le groupe amino N6 des adénines dans le grand sillon de l’ADN.
Les résultats structuraux combinés, de l’AFM, de la cryo-EM, et de la diffusion de neutrons (SANS et INS), permettent de proposer un modèle cohérent du complexe fibrillaire formé par Hfq et l’ADN. Ils révèlent un nouveau mécanisme de pontage de l’ADN par protéine amyloïde ouvrant une nouvelle compréhension de la mise en forme du génome et de la régulation de l’expression des gènes chez les procaryotes. Ce mécanisme pourrait également s’appliquer aux eucaryotes, où des amyloïdes ont été découverts. Ces résultats remettent ainsi en question la compréhension traditionnelle de la manière dont les protéines, peuvent organiser et compacter l’ADN au moyen d’un domaine amyloïde.
Références :
[1] « Amyloid-like DNA bridging: a new mode of DNA shaping. »
Wien, F., M. Gragera, T. Matsuo, G. Moroy, M. T. Bueno-Carrasco, R. Arranz, A. Cossa, A. Martel, H. N. Bordallo, S. Rudic, M. Velez, J. R. C. van der Maarel, J. Peters and V. Arluison, Nucleic Acids Res 53(5) (2025) gkaf169. https://doi.org/10.1093/nar/gkaf169.
[2] « Cryo soft X-ray tomography to explore Escherichia coli nucleoid remodeling by Hfq master regulator. »
Cossa, A., S. Trepout, F. Wien, J. Groen, E. Le Brun, F. Turbant, L. Besse, E. Pereiro and V. Arluison. J Struct Biol 214(4) (2022) 107912. doi: https://doi.org/10.1016/j.jsb.2022.107912
Voir aussi :
- Ce travail a fait l’objet d’une actualité scientifique de Science and Technology Facilities Council/ ISIS – Neutron and muon source : « A new way of folding DNA« .
- Voir également l’actualité de l’Université Paris-Saclay : « Nouveau mode de compaction de l’ADN identifié avec l’apport de la diffusion de neutrons« .
Contact CEA-IRAMIS : Véronique Arluison (LLB/MMB).
Collaboration :
- Frank Wien : Synchrotron Soleil, Saint-Aubin 91192 Gif-sur-Yvette Cedex, France
- Judith Peters : LiPhy, Université Grenoble Alpes – CNRS, 38000 Grenoble, France
- Marcos Gragera : Centro Nacional de Biotecnología (CNB-CSIC) Madrid, Spain
- Anne Martel : Institut Laue-Langevin, 38042 Grenoble Cedex 9, France
- Tatsuhito Matsuo : Hiroshima International University (HIU), Hiroshima, 739-2695, Japan
- Gautier Moroy : Université Paris Cité, CNRS, INSERM, F-75013 Paris, France
- Heloisa N. Bordallo : The Niels Bohr Institute, University of Copenhagen, DK-2100 Copenhagen, Denmark
- Svemir Rudic : ISIS Neutron and Muon Source, SFTC, Rutherford Appleton Laboratory, Didcot, OX11 0QX, United Kingdom
- Johan van der Maarel, National University of Singapore, Singapore