Pour survivre dans leur milieu, les bactéries doivent s'adapter à des conditions de croissance très différentes. Cette propriété est de première importance lorsque ces dernières doivent coloniser un hôte. Bien que la façon dont elles s'adaptent à leur milieu ne soit pas encore complètement comprise, il a récemment pu être montré que les bactéries utilisent pour cela une protéine appelée Hfq et des acides ribonucléiques (ARN) qui lui sont associés, les deux ayant la propriété de s’auto-assembler en nanostructures. Cette thématique est l’une de celles développées au sein de l’IRAMIS.
Une équipe de l’IRAMIS/LLB associée à des équipes de l’I2BC, du Synchrotron SOLEIL et de l’Institut Curie d’Orsay, s’est intéressée à une nouvelle propriété des assemblages formés par Hfq : ces équipes ont pu montrer que la protéine bactérienne forme des structures amyloïdes, comme celles retrouvées lors de maladies neuro-dégénératives, et que ces structures protéiques affectent l’intégrité des membranes biologiques. Ce résultat publié dans la revue Scientific Reports pourrait avoir d’importantes conséquences pour comprendre la communication bactérienne et pourrait être utilisé pour développer de nouveaux agents antibactériens.

Les bactéries sont des organismes unicellulaires qui doivent constamment s'adapter aux changements du milieu extérieur. Cette propriété est particulièrement critique pour les bactéries pathogènes qui doivent ajuster leur métabolisme en fonction des conditions de croissances retrouvées dans l'hôte. Pour se faire, elles ont donc du développer des moyens pour changer rapidement leur expression génétique et ont du apprendre à communiquer entre elles. Un des moyens utilisé pour cela fait intervenir une protéine appelée Hfq et des acides ribonucléiques de petite taille ne codant pour aucune protéine (appelés ARN non codants) [1]. Ces deux partenaires permettent à la bactérie de changer rapidement son expression génétique, par exemple lors d’une carence en fer.
L’étude récente menée par une équipe de l’IRAMIS/LLB associée à des équipes de l’I2BC, du synchrotron Soleil et de l’institut Curie d’Orsay a permis de montrer que les deux partenaires (Hfq et ARN) ont la propriété de s’assembler en nanostructures [2,3]. Ils ont notamment pu montrer que Hfq forme des assemblages de types amyloïdes, comme ceux retrouvés dans des pathologies neuro-dégénératives [4]. Cependant dans ce cas précis, les assemblages formés ont une fonction servant la physiologie de la cellule et sont appelés amyloïdes fonctionnels. Différentes approches de biophysique ont été menées en vue de caractériser ces nanostructures (notamment par diffusion aux petits angles de rayons X (SAXS) ou de neutrons (SANS), microscopie moléculaire TEM/AFM et spectroscopie SRDC/fluorescence).
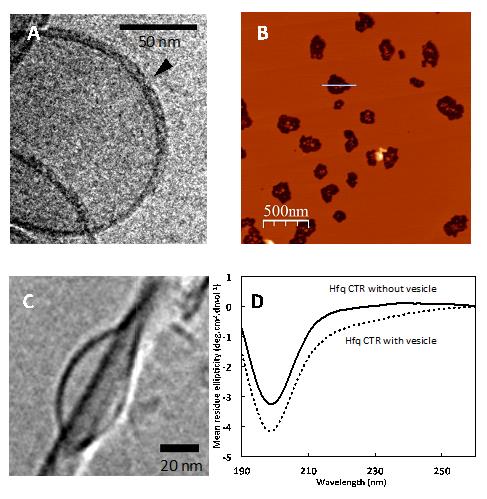
(B) Visualisation par Microscopie de Force Atomique (AFM) de la formation de pores dans la membrane suite à la fixation de la protéine Hfq, résultant en l'éclatement de la vésicule initiale de 200 nm, en petites vésicules d'environ 20 nm
(C) Vue en cryo-microscopie electronique en transmission (Cryo-TEM *) d'une fibre amyloïde en interaction avec un liposome (« bulle » allongée).
(D) Spectre en dichroïsme circulaire (avec rayonnement synchrotron) montrant le changement de structure secondaire de Hfq suite à sa fixation sur la vésicule.
© Creative Common : figure adaptée de Malabirade et al Scientific reports 7: 10724.
Très récemment, ces équipes ont de plus pu mettre à jour une nouvelle propriété des assemblages de Hfq: leur capacité à interagir avec les membranes et à en affecter leur intégrité. Ces travaux publiés dans la revue Scientific reports [5] ouvrent de nouvelles perspectives sur les moyens de communication utilisés par les bactéries. En effet, les bactéries utilisent en général de petites molécules pour communiquer entre elles (système dit du « quorum sensing ») [6]. Mais ces nouveaux travaux permettent de penser que Hfq pourrait permettre l’export d’acides nucléiques entiers hors de la cellule en formant des pores transitoires dans la membrane bactérienne. Ceci pourrait permettre l’échange direct de matériel génétique dans le milieu extracellulaire. Bien que le mécanisme moléculaires ne soit pas encore totalement mis à jour, cette hypothèse semble cependant plausible compte tenu de l’identification d’ARN non codants bactériens dans le milieu extracellulaire [7]. Cette nouvelle voie de régulation pourrait être utilisée pour développer de nouveaux antibiotiques.
* Cryo-TEM : procédé d'imagerie par microscopie électronique à transmission après congélation, préservant la morphologie et la structure de molécules biologiques. Les chercheurs à l'origine de cette technique ont été récompensés par le Prix Nobel de Chimie 2017.
Références
[1] Hfq and its constellation of RNA
J. Vogel & B.F. Luisi, Nat Rev Microbiol 9, 578-589 (2011)
[2] Multiple approaches for the investigation of bacterial small regulatory RNAs self-assembly
C. Lavelle, F. Busi, & V. Arluison, Methods Mol Biol 1297, 21-42 (2015).
[3] Techniques to analyse sRNA protein cofactor self-assembly in vitro »
D. Partouche, A. Malabirade, T. Bizien, M. Velez, S. Trepout, S. Marco, V. Militello, C. Sandt, F. Wien and V. Arluison, Methods Mol Biol (2018),
in « Bacterial regulatory RNA methods in molecular biology », Editors: Veronique Arluison & Claudio Valverde, SPRINGER (2018) .
[4] New insight into the structure and function of Hfq C-terminus
E. Fortas, F. Piccirilli, A. Malabirade, V. Militello, S. Trépout, S. Marco, A. Taghbalout A, V. Arluison Biosci Rep 35 (2015).
[5] Membrane association of the bacterial riboregulator Hfq and functional perspectives
A. Malabirade, J. Morgado-Brajones, S. Trépout, F. Wien, I. Marquez, J. Seguin, S. Marco, M. Velez & V. Arluison. Sci Rep 7, 10724 (2017).
[6] Comprehensive analysis reveals how single nucleotides contribute to noncoding RNA function in bacterial quorum sensing
S.T. Rutherford, J.S. Valastyan, T. Taillefumier, N.S. Wingreen & B.L. Bassler
Proc Natl Acad Sci U S A 112, E6038-6047.
[7] The extracellular RNA complement of Escherichia coli
Ghosal A, B.B. Upadhyaya, J.V. Fritz, A. Heintz-Buschart, M.S. Desai, D. Yusuf, D. Huang, A. Baumuratov, K. Wang , D. Galas, P. Wilmes, Microbiologyopen (2015).
Voir le fait marquant Synchrotron SOLEIL : Amyloïdes fonctionnels et adaptation bactérienne.
Contact CEA-IRAMIS : Véronique Arluison (LLB)
Collaborations :
- F. Wien, Synchrotron SOLEIL, L’Orme des Merisiers, Saint-Aubin BP 48 91192 Gif-sur-Yvette Cedex, France
- J. Seguin : I2BC, CEA, CNRS, Univ. Paris-Sud, Université Paris-Saclay, 91198, Gif-sur-Yvette, Cedex, France
- S. Trepout : Institut Curie & INSERM U 1196, CNRS UMR 9187, Université Paris Saclay, Université Paris-Sud, Bât 110-112, Centre Universitaire, Rue Henri Becquerel, 91405, Orsay, France
- M. Velez : Instituto de Catálisis y Petroleoquímica, CSIC, c/Marie Curie, 2, Cantoblanco, E-28049, Madrid, Spain


