Trois » métiers » de l'IRAMIS trouvent une extension naturelle vers la biologie :
- L'ingénierie moléculaire, où les études d'interactions coopératives de molécules en solution trouvent une suite directe dans l'étude des protéines et des différents modes d'assemblage de molécules d'intérêt biologique,
- L'étude de la matière à haute densité d'énergie, où les travaux sur la radiolyse et les interactions rayonnement-molécule, se transposent directement à des molécules comme l'ADN,
- L'étude de la matière ultra divisée, domaine dans lequel les matériaux nanostructurés, la nanophysique et la biologie convergent naturellement.
L'utilisation de techniques et de savoir faire qui étaient traditionnellement du domaine du physicien ou du chimiste, pour l'étude d'objets biologiques se révèle fructueuse. Celà concerne en particulier les neutrons et les méthodes de RMN non conventionnelle spécifiquement développées pour l'étude des protéines. Les analyses ICP-MS (couplage torche à plasma – spectrométrie de masse) et par microsonde nucléaire pour l'étude d'échantillons biologiques (micro-organismes, cellules, tissus, plantes …) et les études environnementales en sont d'autres exemples.
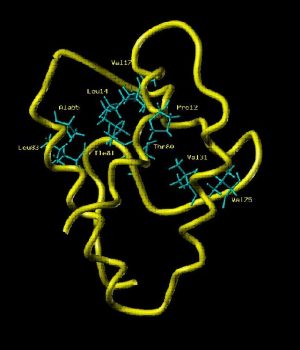
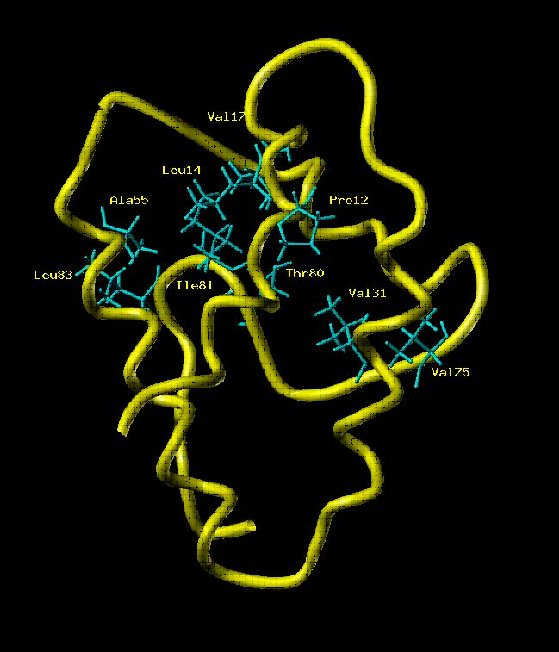
← Squelette peptidique de la LTP, Lipid Transport Protein, de 90 résidus (RMN avec Xénon polarisé par laser). (P. Berthault NIMBE)
Three « topics » of IRAMIS have found natural extensions towards biology:
- Molecular engineering, where studies of cooperative interactions of molecules in solution are directly related to those on proteins as well as on the various modes of assembly of molecules of biological interest,
- Matter with high energy density, like radiolysis and studies of radiation molecule interactions, which transpose directly to molecules like DNA,
- Ultra-divided matter, where studies of nanostructured materials, nanophysics and biology converge naturally.
The use of techniques and know-how, traditionally in the domain of the physicist or chemist, for the study of biological objects proves to be fruitful. This applies in particular to neutron diffusion studies and unconventional NMR methods specifically developed for the study of proteins. ICP-MS (plasma torch-mass spectrometry) and nuclear microprobe analyzes for the study of biological samples (Micro-organisms, cells, tissues, plants …) and environmental studies are further examples.