Molécules clés du vivant, les polymères d'acides nucléiques peuvent se présenter sous la forme d'une chaine moléculaire unique ou sous une forme multichaines (tel que la double hélice de l'ADN), qui adoptent une grande variété de conformations, pouvant comprendre des hélices simples ou doubles, des boucles, des pseudo-nœuds des structures en duplexes, triplexes ou quadruplexes. Ces structures ont toutes des rôles cruciaux et bien spécifiques en biologie, et leur rôle va de la réplication de l’ADN en vue de la transmission de l’information génétique, à la régulation de l'expression des gènes, en passant par le contrôle de la stabilité du génome.
Afin d'améliorer l'analyse de la conformation de ces macromolécules, une nouvelle base de données a été créée dans le cadre d'une approche scientifique concertée : des chercheurs de Pologne, de France et des États-Unis ont uni leurs efforts pour collecter et normaliser les spectres de dichroïsme circulaire (CD) d’acides nucléiques, ADN et ARN. Plus de 150 spectres, tirés de la littérature, ou récemment obtenus sur la ligne de lumière DISCO, ont été rassemblés dans cette base structurée « NACDDB – Nucleic Acid Circular Dichroism Database », et rendus ainsi accessibles au public le plus large.
Les acides nucléiques tels que l'ADN, connu pour être le support de l’information génétique, et l'ARNm, récemment utilisé pour certains vaccins contre le COVID-19, sont des macromolécules constituées d'un squelette de phosphate (hélice), de sucres (oxy ou désoxy-ribose) et de 5 molécules azotées appelées « bases » : purines et pyrimidines, notées A, C, G, T et U. Leur structure moléculaire et leur arrangement spatial, comme l'hélice à double brin pour l’ADN, est cruciale pour leur fonction.
La spectroscopie de dichroïsme circulaire mesure la différence d’absorption de la lumière circulaire polarisée droite et gauche dans le domaine de l'ultraviolet par des molécules chirales*, comme le sont les acides nucléiques par exemple. Dans ce domaine de rayonnement – situé, en termes d’énergie, entre 5 et 7 eV – les électrons des polynucléotides sont excités par la lumière et sont à l'origine de l'absorption UV. Les bandes d'absorption caractéristiques de la lumière UV polarisée circulairement (droite ou gauche) dans les sous-domaines UV communément appelés UV A, B et C (soit, en termes de longueurs d’onde, entre 170 et 320 nm) permettent de distinguer différents types de repliement de ces macromolécules complexes. Leur arrangement spatial peut être corrélé avec les spectres CD, ce qui aide à déterminer le repliement des acides nucléiques étudiés.
Au cours de ces deux dernières décennies, les biologistes structuraux ont collecté, enregistré et passé au crible les spectres CD obtenus sur des protéines, ce qui leur a permis de déterminer avec succès les structures de repliement dites secondaires (hélices α, feuillets β et pelotes aléatoires) pour des protéines amyloïdes, des protéines membranaires et des complexes protéine-protéine notamment.
Une des différences entre les acides nucléiques et les protéines réside dans la taille et la position de leur chromophore, molécule responsable de leurs propriétés chirales : pour les protéines, le chromophore principal est la liaison peptidique de petite taille reliant de manière covalente chacun des acides aminés constituant la protéine. Pour les acides nucléiques, c’est le nucléotide constitué d’un phosphate, un sucre et une base qui forment le chromophore ; or il est plus grand et les combinaisons possibles sont plus variées que la liaison peptidique. La multitude de distances observées, par exemple chez les acides nucléiques à simple ou double brin, laisse beaucoup de possibilités pour l'attribution d'excitations électroniques.
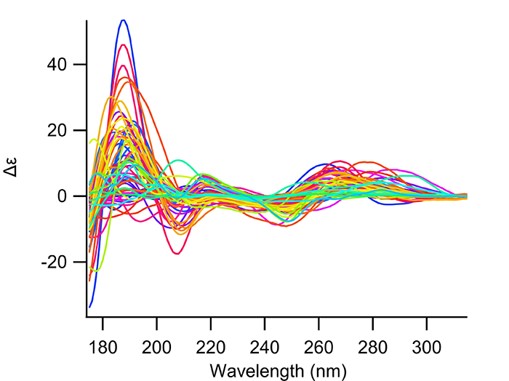
Les scientifiques ont alors collectés les spectres CD d'acides nucléiques publiés depuis la fin des années 50. Dans un premier temps ils ont systématiquement numérisé les près de 90 spectres issus dans la littérature, auxquels ont été ajoutés des données expérimentales récemment collectées sur la ligne de lumière DISCO à SOLEIL, avec 63 spectres de dichroïsme circulaire de rayonnement synchrotron (SRCD) sur des macromolécules d'ADN et d'ARN communes et spéciales (comme des nucléotides méthylés) en solution (figure 1).
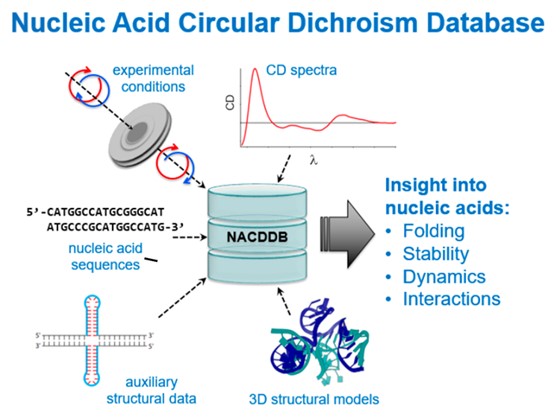
L'ensemble de ces spectres (plus de 150) ont été déposés dans la base NACDDB – Nucleic Acid Circular Dichroism Database, mise librement à disposition avec des métadonnées portant sur les conditions expérimentales comme le pH, la salinité et la température), les modèles 3D et les liens vers les références bibliographiques correspondantes. Les spectres de différents types de molécules d'acide nucléique ont ainsi été déposés : ADN, ARN, complexes de ces deux types d’acides nucléiques, mais aussi des dérivés d'acides nucléiques modifiés présentant un intérêt pharmaceutique.
La base de données permet la recherche, la comparaison, le téléchargement de spectres CD et sera d'une aide précieuse pour permettre la détermination des arrangements structuraux de macromolécules à base d'acides nucléiques. Pour les séquences dont les structures 3D à haute résolution ont déjà été déterminées, ces structures sont également disponibles dans la base. La NACDDB est aussi ouverte à la soumission de nouveaux spectres CD d'acides nucléiques, obtenus dans des laboratoires de biologie structurale sur des spectrophotomètres de pointe ou sur des lignes de lumière synchrotron mettant en œuvre la technique de SRCD.
*Chiral (mot grec signifiant « main ») : propriété de symétrie d'un objet qui n'est pas superposable à son image dans un miroir (comme une main droite, dont le reflet est une main gauche…). Les molécules chirales contiennent des centres chiraux comme les carbones tétraédriques avec quatre substituants différents, ou comme les atomes de carbone impliqués dans les liaisons peptidiques des protéines. La présence de centres chiraux et leur arrangement local peuvent être sondés avec de la lumière polarisée circulairement.
Références :
F. « NACDDB: Nucleic Acid Circular Dichroism Database »,
A. Cappannini, K. Mosca, S. Mukherjee, S.N. Moafinejad, R.R. Sinden, V. Arluison, J. Bujnicki, F. Wien, Nucleic Acids Research (2022) gkac829.
Voir l'actualité du Synchrotron Soleil : « NACDDB : Une base de données de dichroïsme circulaire des acides nucléiques » / NACDDB: Nucleic Acid Circular Dichroism DataBase.
Contacts :
- Synchrotron Soleil, ligne DISCO : Frank Wien,
- LLB, UMR CEA-CNRS : Véronique Arluison, LLB/MMB.
Collaboration :
- Laboratoire Léon Brillouin, LLB – UMR 12 CEA-CNRS, Centre CEA Saclay, 91190 Gif-sur-Yvette
- UFR Sciences du Vivant, Université Paris Cité
- International Institute of Molecular and Cell Biology, University of Warsaw
- South Dakota School of Mines and Technology (USA)