Cette étude propose une méthode innovante de détection de protéines intracellulaires qui associe fluorescence et résonance magnétique, en combinant l’utilisation d’un fluorophore activable de très petite taille et l’exploitation de la grande sensibilité d’un traceur RMN non toxique, le xénon, dont le spin nucléaire est hyperpolarisé. Les biosondes ainsi constituées sont ainsi doublement activables, combinant un signal de fluorescence et un signal de RMN du xénon-129 spécifiques lorsque la cible est rencontrée.

La mise en évidence et l’examen de mécanismes biologiques intracellulaires nécessitent des techniques sensibles et non invasives. L’utilisation combinée de plusieurs méthodes d’analyse permet de dépasser les limites de chacune d’elles et d’améliorer leur spécificité. C’est le point de vue des approches multimodales, très développées ces dernières années, en particulier pour l’imagerie moléculaire in vivo. Les techniques d’imagerie par fluorescence sont faciles à mettre en œuvre et largement utilisées en raison de leur sensibilité élevée et de leur excellente résolution spatiale et temporelle, mais elles présentent généralement une faible spécificité, puisqu’il suffit que le motif peptidique de la protéine cible soit présent pour modifier le signal de fluorescence. Le signal de résonance magnétique nucléaire (RMN) peut combler ce manque, à condition que l’on pallie sa faible sensibilité.
Dans cette étude, des molécules-cages, encapsulant de manière réversible le xénon, et auxquelles a été adjoint un petit fluorophore, ont été conçues et synthétisées (voir figure ci-contre) [1].
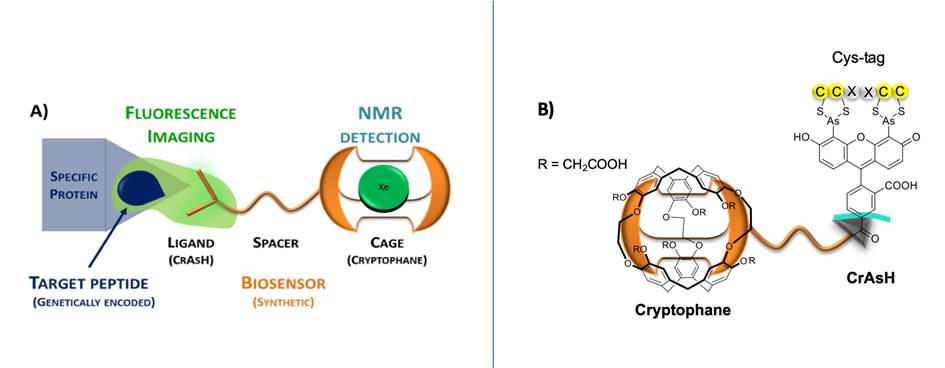
Pour la partie fluorescence, le groupement fluorogène arsénié (CrAsH, sonde d’affinité multi-usage à fluorescence non spécifique) peut se lier au motif peptidique Cystéine-Cystéine-X-X-Cystéine-Cystéine (où X est n’importe quel acide aminé). Son intensité de fluorescence est alors exaltée de plusieurs ordres de grandeur [2]. Avec sa petite taille et tel qu’il est fixé, la présence du fluorophore n’altère pas la structure – donc l’activité – de la protéine cible.
Pour la partie résonance magnétique, le xénon, dont la polarisation nucléaire – donc le signal RMN – a été augmentée de plusieurs ordres de grandeur par pompage optique (hyperpolarisation) est initialement introduit de façon indépendante. Le gaz est spontanément encapsulé de manière réversible dans la molécule-cage faisant partie de la biosonde, et donne alors une signature spectrale originale. Celle-ci est encore modifiée lorsque la biosonde rencontre sa cible. Cette fréquence de résonance étant très spécifique, seuls les noyaux de Xe dans cette situation sont sondés et perdent leur polarisation. Le renouvellement continu du Xe dans les molécules cages permet de répéter la détection, ce qui peut apporter un gain de sensibilité ou la possibilité d’un suivi cinétique. Ainsi, côté RMN, l’intérêt de l’utilisation du Xe polarisé est multiple :
- un fort gain en signal apporté par l’hyperpolarisation,
- le xénon franchissant la barrière cellulaire sans perdre son hyperpolarisation peut être introduit a posteriori de manière décorrélée de l’administration de la biosonde,
- la grande sensibilité du xénon à son environnement local permet d’avoir une signature spectrale univoque de l’interaction de la biosonde avec la protéine.
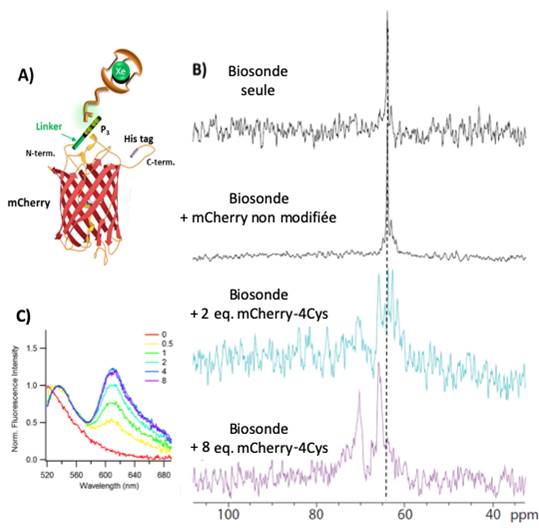
Les mesures apportant la preuve de concept de cette approche ont été réalisées en utilisant des protéines fluorescentes dans le rouge (mCherry) contenant le motif Cys-Cys-X-X-Cys-Cys reconnu par la biosonde, produites par des bactéries génétiquement modifiées.
Ce travail se poursuit par des études in cellulo. Il est à noter que dans cette approche RMN utilisant le xénon ainsi vectorisé, le gaz rare peut être administré de nombreuses fois avant et après apparition du complexe biosonde – protéine, ce qui permet un suivi longitudinal que la fluorescence seule ne permettrait pas facilement ; le xénon est la seule espèce hyperpolarisée offrant cette possibilité.
Références :
[1] “Bimodal detection of proteins by 129Xe NMR and fluorescence spectroscopy”
E. Mari, Y. Bousmah, C. Boutin, E. Léonce, G. Milanole, T. Brotin, Patrick Berthault, M. Erard, ChemBioChem 20 (2019) 1.
[2] “A doubly responsive probe for the detection of Cys4-tagged proteins“
N. Kotera, E. Dubost, G. Milanole, E. Doris, E. Gravel, N. Arhel, T. Brotin, J.-P. Dutasta, J. Cochrane, E. Mari, C. Boutin, E. Léonce, P. Berthault, B. Rousseau, Chemical Communications 51 (2015) 11482.
Contact CEA-IRAMIS : Patrick Berthault (NIMBE/LSDRM)
Collaboration :
- M. Erard, Y. Bousmah : Laboratoire de Chimie Physique, Université Paris Sud, Université Paris-Saclay, Bat 349, 91405 Orsay, France
- T. Brotin : Laboratoire de Chimie, Ecole Normale Supérieure de Lyon, CNRS UMR 5182, Université Lyon 1, 46 allée d’Italie, 69364 Lyon, France
- G. Milanole : SCBM, Université Paris-Saclay, CEA Saclay, 91191 Gif-sur-Yvette, France.





