Domaine, spécialité : Biochimie, Biophysique, Biologie structurale
Mots-clés : protéines membranaires, TSPO, systèmes d’expression, spectroscopies optiques, diffusion de rayons X et de neutrons (SAXS/SANS)
Unité d’accueil : LLB/MMB
Résumé
Ce stage offre l’opportunité d’explorer TSPO, une protéine membranaire mitochondriale intrigante, utilisée en neuro-imagerie comme marqueur de l’inflammation cérébrale dans les cancers et maladies neurodégénératives. L’objectif est de produire TSPO sous forme native puis d’analyser sa structure et son fonctionnement grâce à des approches biophysiques et structurales de pointe (spectroscopies optiques, diffusion de rayons X et de neutrons, thermophorèse). Ces recherches permettront de mieux cerner le rôle de TSPO et de contribuer au développement de nouveaux outils d’imagerie et de thérapie.
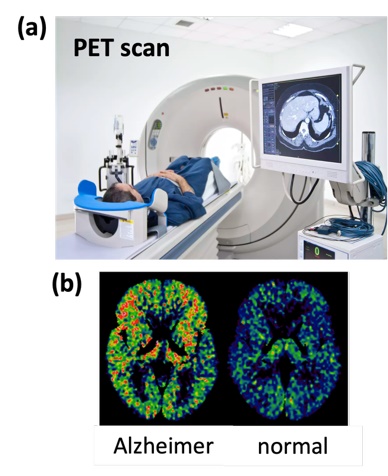
Fig. 1. TSPO comme cible thérapeutique en neuro-imagerie.
(a) tomographie par émission de positons (PET) ; (b) surexpression de TSPO dans le cerveau d’un patient atteint de la maladie d’Alzheimer
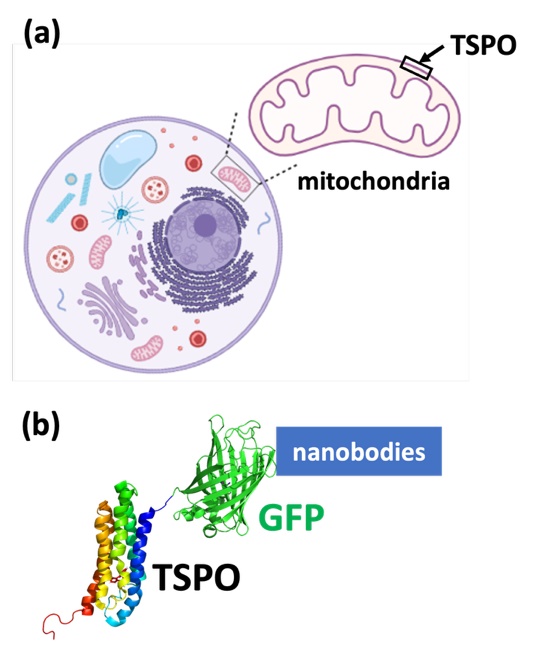
Fig. 2. Production et purification de TSPO. (a) Localisation de TSPO dans la membrane externe de la mitochondrie ; (b) stratégie de purification basée sur la reconnaissance de la protéine de fusion fluorescente GFP par des « nanobodies ».
Sujet détaillé
👉 Objectif 1 : Produire TSPO sous forme native
À l’aide de la plateforme ProtEx (I2BC, Saclay), nous avons déjà établi une preuve de concept en exprimant TSPO en conditions natives dans la levure S. cerevisiae. Le stage consistera à optimiser ce protocole de production et purification, notamment dans différents environnements (détergents, mélanges détergents/lipides, nanodisques lipidiques).
👉 Objectif 2 : Explorer sa structure et son fonctionnement
La protéine produite sera caractérisée par un panel de techniques biophysiques et structurales :
- spectroscopies optiques (absorbance, fluorescence, dichroïsme circulaire),
- diffusion de lumière (MALS, DLS/SLS),
- diffusion aux petits angles des rayons X et neutrons (SAXS au synchrotron SOLEIL, SANS à l’ILL Grenoble),
- mesures d’affinité pour des ligands par thermophorèse (MST).
Les données obtenues permettront de comparer la forme APO et la forme liée à un ligand, pour mieux comprendre les mécanismes de fixation et de stabilité.
Références :
- Structure/function of mTSPO translocator in lipid :surfactant mixed micelles,
Saade C, Pozza A, Bonnete F, Finet S, Lutz-Bueno V, Tully MD, Varela PF, Lacapere JJ, Combet S. Enhanced, Biochimie 224, 3, 2024. - Effect of amphiphilic environment on the solution structure of mouse TSPO translocator protein,
Combet S, Bonneté F, Finet S, Pozza A, Saade C, Martel A, Koutsioubas A, Lacapère JJ. Biochimie 205, 61-72, 2023.
Lieu du stage
Laboratoire Léon-Brillouin (CEA, CNRS, Université Paris-Saclay), CEA-Saclay, en collaboration avec l’I2BC/B3S, Gif-sur-Yvette, et l’IBPC, Paris.
Conditions de stage
- Durée du stage : 4 à 6 mois
- Niveau d’étude requis : Bac+5
- Formation : Master 2, école d’ingénieurs
- Poursuite possible en thèse : oui
- Date limite de candidature : 30 janvier 2025
Compétences requises
Étudiant de M2 en biophysique, biochimie ou physico-chimie, curieux et motivé par la biologie structurale et les protéines membranaires.
Langue : français ou anglais
Liens utiles
- Site web du laboratoire : https://iramis.cea.fr/llb/mmb
- Site web de l’encadrant : https://iramis.cea.fr/en/pisp/sophie-combet/
- Fiche détaillée du sujet de stage
Responsable du stage
Sophie COMBET
Tél. : +33 1 69 08 67 20
E-mail :




