Groupes de recherche / Laboratoires
-

INFRA
Thèmes de recherche Développement de l’instrumentation. Techniques et Instruments Neutrons : Sources, détecteurs, spectromètres… and much…
-

MMB – Matière Molle et Biologie
Thèmes de recherche Thèmes de recherche du LLB/MMB Techniques et Instruments Sources de neutrons, Lignes instrumentales,…
-
NFMQ – Nouvelles Frontières dans les Matériaux Quantiques
À la Une Magnétisme multi-échelle Magnétisme quantique Electrons fortement corrélés Matériaux fonctionnels ICONE Outstations Publications récentes…
Instruments diffusion de neutrons
Le LLB conçoit, développe et pilote des instruments d’études par diffusion de neutrons pour la communauté scientifique française et internationale, implantés autour des sources européennes de neutrons. Il mène également des recherches pour concevoir une source compacte de neutrons, à même de remplacer les lignes de neutrons issues de réacteur nucléaire de recherche.
À la Une
-
Prix ENSA 2025 à Dalila Bounoua, Iurii Kibalin et Juan Rodriguez-Carvajal
Lors de l’International Conference on Neutron Scattering (ICNS) 2025 – 6-10 Juillet à Copenhague (Danemark) et Lund (Suède), remise de plusieurs prix de l’ENSA – European Neutron Scattering Association à des chercheurs travaillant ou qui ont travaillé au LLB – Laboratoire Léon Brillouin : Dalila Bounoua, Iurii Kibalin et Juan Rodriguez-Carvajal.

Agenda
-
ILL call for proposal : 15/09/2025
Le 15/09/2025 -
Appel à proposition d’expérience sur les appareils de la 2FDN
Le 15/10/202500h34-23h58
Faits marquants scientifiques
-
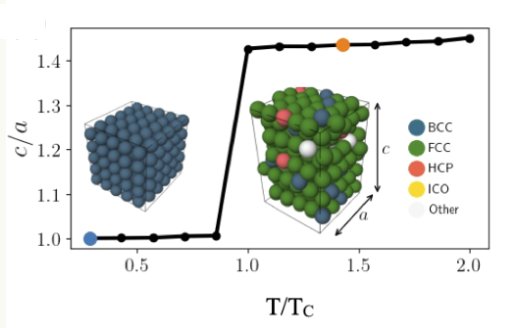
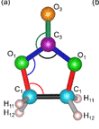
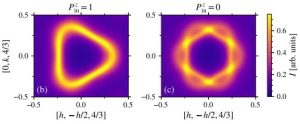
Magnetism in iron alloys: methodological advances for thermodynamics, defects, and kinetics
Steels are among the technologically and economically most relevant materials. Key innovations in important sectors of human society such as mobility, energy and safety, are currently based on alloying of Fe with other transition-metal elements such as Mn, Cr, or Co.
Publications récentes
Retrouvez les publications scientifiques de nos chercheurs.
Découvrir nos projets
Projets scientifiques du LLB
Nos offres d’emplois
Postes CDD, CDI, Post-doc…
Sujets de stage M2
Thèses, doctorats…
Propositions de sujets de thèse